使用主响应曲线方法(principal response curve analysis)分析刺槐数据
老王 / 2015-01-22
说起来,主响应曲线分析已经帮了自己2次了。一次是发表了一篇SCI论文,即Using microbial community functioning as the complementary environmental condition indicator,用来分析了BIOLOG数据,另一次就是在窄冠刺槐的课题中用了这种方法,增强了点科技感。搜了搜似乎网上还没有针对生态学分析中利用这种方法的中文的介绍,这里简单写一下,当作自己网站的第一篇原创文章。
一、分析目标
针对多元数据,且在一个时间段内有连续的记录,分析在这个过程中有整体什么差异,且能展现这些差异是由哪些指标主要引起的。 这么说有点抽象,举个例子吧。比如有6个不同刺槐品种,从种下来开始,就测量胸径、树高、郁闭度、枝下高等指标,每年测量,一直测量了10年。不同品种肯定有差异,但这些差异在这10年里是怎么变化的呢?当然我可以单独拿出一个指标来,比如胸径,比较十年的变化,但这种比较不具有整体性。为了从整体上把不同品种的生长特征在这10年间的变化刻画出来,就可以使用PRC方法。它其实是一种基于降维的方法,在刻画出每个时间节点的主要特征后,在时间序列上表现出来。出处是Principal Response Curves: Analysis of Time-dependent Multivariate Responses of a Biological Community to Stress.van den Brink, P. J. & ter Braak, C. J. F. (1999)。
二、数据准备
数据准备,行为case,列为variable。即行是测定对象,列是指标。
cihuai = read.table (file=file.path("data for analysis", "PRC analysis data.txt"), header = T, sep="\t", fileEncoding="UTF-16")
# 注意这种选择文件相对路径的方法。chuck中的工作目录和console中的工作目录不同。
head(cihuai)## X dens hight DBH Crown UBhigh Closure
## 1 c1-1-1 833 1.55 1.29 0.52 0.39 0.05
## 2 c1-1-2 833 1.59 1.31 0.53 0.40 0.05
## 3 c1-1-3 833 1.61 1.32 0.54 0.41 0.05
## 4 c2-1-1 833 1.34 1.17 0.69 0.31 0.06
## 5 c2-1-2 833 1.34 1.16 0.68 0.30 0.06
## 6 c2-1-3 833 1.35 1.15 0.68 0.30 0.06上面是从R中数据准备。要注意R中的行名不能重复,因此重复测定用1、2、3区别;此外,最少3个重复,否则无法进行分析。
三、分析过程
分析在R语言中进行,需要用到vegan包。
library(vegan)## Loading required package: permute## Loading required package: lattice## This is vegan 2.5-5?prc #查看prc用法可以知道PRC分析的作用、操作方法和实例。看例子是个好方法,很多抽象的东西看了也不大容易理解,比着葫芦画瓢还是要容易点。
主要函数参数有3个:
prc(response, treatment, time, …)
其中response就是前面说的要准备的数据,treatment是对数据进行的不同处理。如我们刺槐课题,为了比较不同造林模型、不同品种的生长差异,treatment就是不同的造林模型和品种。time是时间,我们这里就是不同的年份。
treatment和time都是用来表示response中的case属于哪种处理,哪个时间点的。如我们刺槐课题,6个处理,每个处理3个重复,18行,6年时间,108行,108个case。treatment和time是这样对应的:
year = gl(6, 18, labels = c(1, 2, 3, 4, 5, 6))
model = factor(rep(c(1,1,1,2,2,2,3,3,3,4,4,4,5,5,5,6,6,6), 6))
cihuai = cihuai[,c(3,4,5,6,7)]数据准备好以后就可以分析了,很简单的运行一下prc函数即可。 分析结果可以用summary函数查看,并可以作图直观的看。
prc.res = prc(cihuai, model, year)
summary(prc.res)##
## Call:
## prc(response = cihuai, treatment = model, time = year)
## Species scores:
## hight DBH Crown UBhigh Closure
## -1.67534 -1.44184 1.72381 -0.12213 0.09386
##
## Coefficients for model + year:model interaction
## which are contrasts to model 1
## rows are model, columns are year
## 1 2 3 4 5 6
## 2 0.026084 0.049591 0.094548 0.110082 0.19789 0.15722
## 3 -0.007850 -0.002553 0.004724 -0.005944 -0.01631 -0.05913
## 4 0.022388 0.051586 0.097456 0.121335 0.20814 0.08494
## 5 -0.001511 -0.005113 0.033979 -0.010440 -0.01760 -0.01863
## 6 0.025004 0.054720 0.097878 0.134502 0.17600 0.10246plot(prc.res)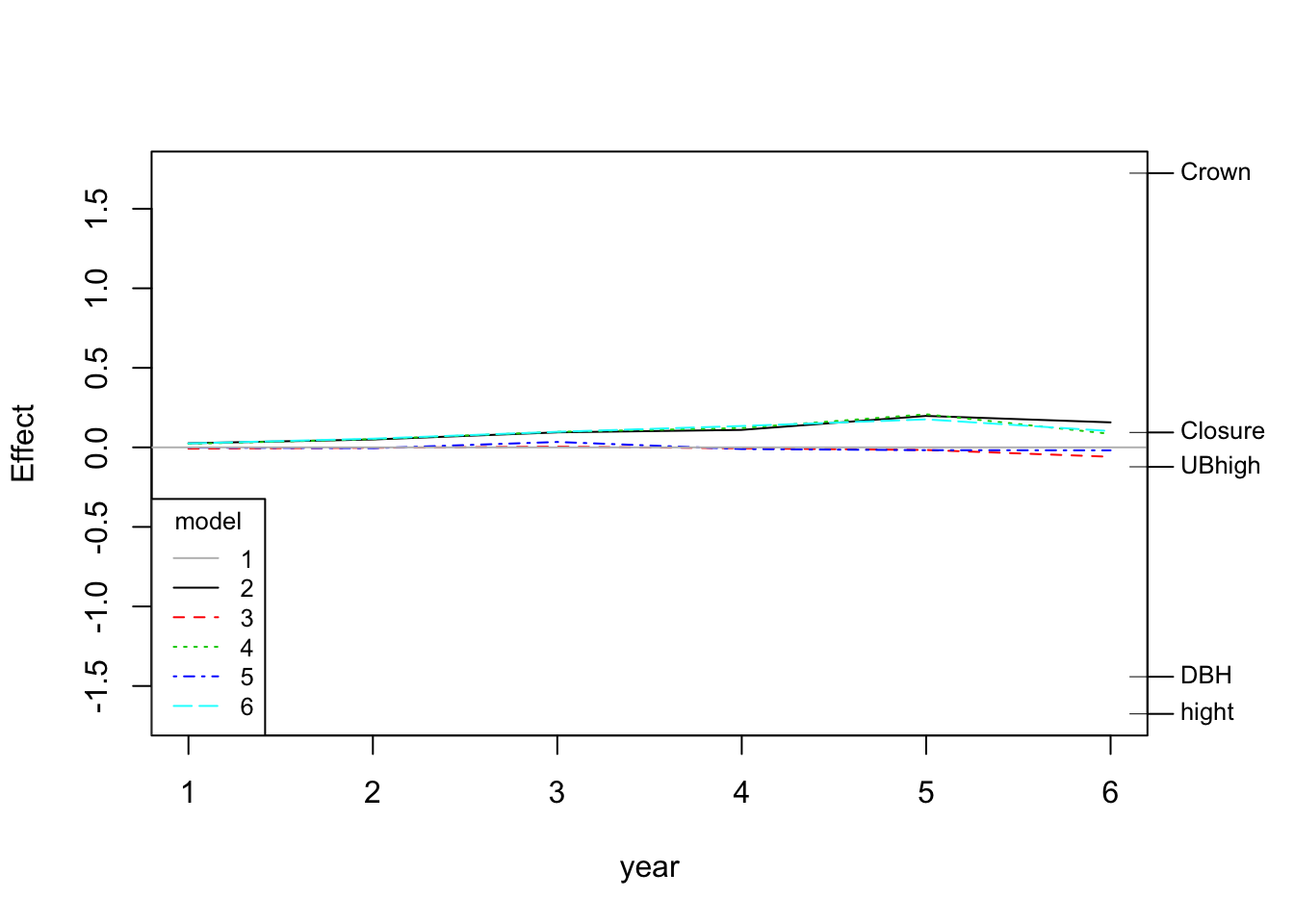
从图上可以发现不同模型在历年来有什么差异,并通过对照右边的指标,知道这些差异主要是由什么引起的。
四、注意事项
有时候需要对数据进行中心化和标准化,以使出来的图更清晰美观。